DOI:10.1021/acsomega.0c03902
电纺纳米纤维由于其仿生结构类似于天然细胞外基质而被广泛用作细胞培养基质。然而,由于纳米纤维中的细胞浸润有限,三维(3D)构建细胞基质并不容易实现。本研究开发了一种将纳米纤维部分酶解成由明胶和聚己内酯(PCL)组成的碎片化纳米纤维的方法。随后用不同浓度的氯仿除去同轴碎片的PCL外壳,以控制外壳上剩余的PCL。明胶核层的膨胀和暴露由剩余的PCL外壳控制。当用碎片化纳米纤维培养细胞时,它们会自发地组装在细胞膜片上。结果表明,碎片化纳米纤维上PCL壳层含量对细胞粘附和增殖具有显著影响。
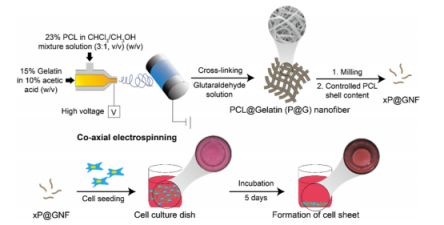
图1.PCL/明胶纳米纤维的制备示意图。使用同轴喷嘴制备了由PCL和明胶组成的核-壳纳米纤维,然后与戊二醛交联(P@G纳米纤维)。将P@G纳米纤维研磨并用氯仿/乙醇的混合物处理,以制备具有部分PCL壳的明胶纳米纤维(xP@GNF,x=(剩余PCL/初始PCL(%,w/w))。用xP@GNF接种细胞以形成细胞片,持续5天。
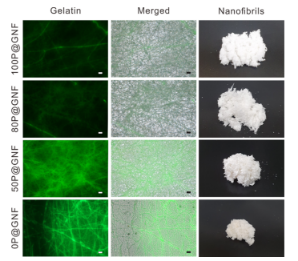
图2.含不同量PCL壳的明胶纳米纤维(GNF)的荧光显微图像。用FITC荧光标记明胶并将其静电纺丝成纳米纤维。在含不同PCL量的表面蚀刻纳米纤维(100P@G纳米纤维,80P@G纳米纤维,50P@G纳米纤维,0P@G纳米纤维)中观察到荧光标记的明胶,与光学显微镜图像重叠的图像(合并),纳米纤维研磨后100P@GNF、80P@GNF、50P@GNF和0P@GNF(10mg)的数字图像(比例尺=10μm)。
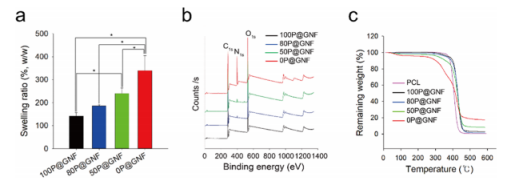
图3.(a)含不同量壳层的P@G纳米纤维的水溶胀率(n=3)。(b)含不同量PCL壳的P@G纳米纤维的XPS光谱。(c)PCL、100P@GNF、80P@GNF、50P@GNF和0P@GNF的热重分析(TGA)。(*)表示统计学显著性(p<0.05),通过单向方差分析(ANOVA)进行评估。
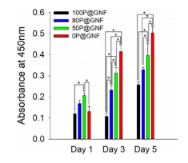
图4.用不同壳含量的P@GNF培养1、3和5天的细胞增殖(n=3)。将NIH3T3细胞与P@GNF混合并接种在未经处理的48孔板中。将WST-1溶液添加到每个孔中,并在黑暗的微生物培养箱中于37℃、5%CO2的条件下孵育1小时。收集上清液,并测量450nm处的吸光度。(*)表示统计学显著性(p<0.05),通过单向方差分析进行评估。
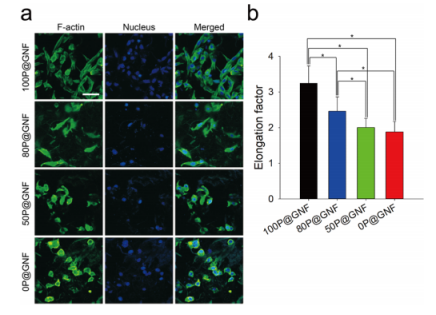
图5.用xP@GNF体外培养NIH3T3细胞。(a)用100P@GNF、80P@GNF、50P@GNF和0P@GNF培养NIH3T3细胞12h,并用4′,6-二氨基-2-苯基吲哚(DAPI)(蓝色,细胞核)和Alexa Fluor 488标记鬼笔环肽(绿色,F-肌动蛋白)染色的共聚焦激光扫描显微图像(比例尺=50μm)。(b)通过以下公式计算伸长率:伸长率=(长轴)/(短轴)。长轴和短轴由ImageJ测量(n>20)。(*)表示统计学显著性(p<0.05),通过单向方差分析进行评估。
